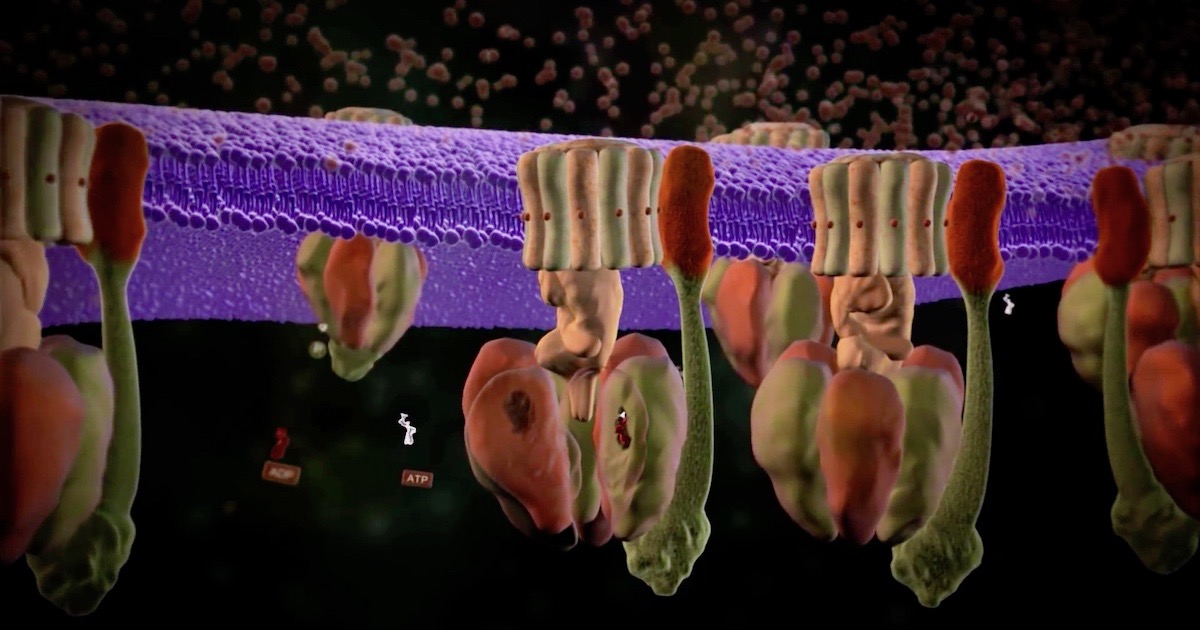
Las imágenes de las máquinas moleculares con las que Michael Behe llamó la atención del público hace 21 años eran tenues y borrosas en ese momento, pero eran lo suficientemente convincentes como para defender una complejidad irreducible. Ahora, las nuevas técnicas de obtención de imágenes, como la microscopía crioelectrónica, permiten a los científicos observar partes individuales de las máquinas con una resolución casi atómica.
ATP sintasa
En 1997, John E. Walker compartió el Premio Nobel de Química con Paul Boyer y Jens Skou por descubrir que el ATP se sintetizaba en las células mediante un motor rotativo. Tenía 56 años en ese momento; ahora, a los 79 años, todavía se dedica a la investigación del mecanismo de la ATP sintasa (ver nuestra animación). Como coautores de un artículo en PNAS1, Walker y otros dos utilizaron microscopía crioelectrónica para examinar el motor a una resolución más alta de la que era posible hace 23 años. El resumen de su artículo presenta noticias sobre esta maravilla giratoria:
La estructura de la ATP sintasa dimérica de las mitocondrias bovinas determinada en tres estados rotacionales por crioscopía electrónica proporciona evidencia de que la captación de protones de la matriz mitocondrial a través del medio canal de entrada de protones procede a través de un mecanismo de Grotthus, y un mecanismo similar puede operar en el salida medio canal. La estructura ha proporcionado información sobre la arquitectura, la constitución mecánica y las propiedades del tallo periférico, parte de la región extrínseca de la membrana del estator, y cómo la acción del tallo periférico amortigua los movimientos de balanceo de lado a lado que ocurren en el complejo de la enzima durante el ciclo catalítico. [Énfasis añadido.]
Un «mecanismo de Grotthus» es una serie de transferencias, algo así como una brigada de baldes. Los protones que alimentan el rotor no fluyen por sí mismos; son pasados a la entrada por una brigada de baldes de moléculas de agua, que toman cada protón y lo entregan a la siguiente molécula de agua.
Varios otros hechos interesantes salieron a la luz en el documento. Como dicen, hay un «movimiento de balanceo de lado a lado» en la máquina. ¡Este motor realmente zumba! Como la porción F1 produce rápidamente moléculas de ATP en un proceso de tres etapas, es inevitable algo de balanceo. El tallo periférico ayuda a amortiguar esos movimientos. Esto ayuda a «aumentar la eficiencia de la enzima» al mantener la subunidad a en su lugar, es decir, la subunidad conectada al carrusel giratorio de F0 en el «dominio de membrana» donde F0 está anclado. Un buen diseñador no querría que las vibraciones sacudan el rotor. El tallo periférico se encarga de eso:
Se debe permitir cierto grado de movimiento del dominio catalítico para permitir la conversión eficiente del par de rotación del tallo central en cambios conformacionales en las subunidades catalíticas, pero sin desperdiciar energía por el desplazamiento innecesario de la porción extrínseca de la enzima. La flexibilidad del tallo periférico mide efectivamente este equilibrio, evitando la rotación libre del dominio catalítico, acomodando los movimientos de balanceo inducidos por el tallo central asimétrico y amortiguando el desplazamiento del dominio de la membrana ayudando a mantener la subunidad a en su lugar.
Sistemas de sintasa
Desde 1997, los investigadores también encontraron que las máquinas de ATP sintasa vienen en pares (dímeros). Los dímeros, además, están montados en filas en los pliegues de las crestas (las membranas dentro de las mitocondrias). El ángulo preciso entre los dos motores dimerizados y el espacio entre ellos maximiza la ingesta de protones. El nuevo artículo dice que el ángulo entre los dímeros se flexiona un poco, amortiguando aún más las vibraciones, gracias a moléculas específicas en el pivote de la cuña que permiten cierta flexión, pero no demasiado:
Nuestra estructura de dímeros bovinos tiene una cuña hecha de pequeñas proteínas y lípidos específicos en el dominio de la membrana de cada monómero que impone un rango de ángulos agudos en los ejes centrales de los monómeros, y un pivote entre las cuñas acomoda los movimientos de balanceo de la máquina acompañando la catálisis y otros movimientos que suceden de forma independiente. También arroja luz sobre cómo se hace girar el rotor de membrana.
Los científicos del Instituto de Ciencia y Tecnología de Austria también han estado trabajando en la ATP sintasa. Se jactan de haber resuelto la estructura de la «turbina más pequeña del mundo» que Leonid Sazanov llama «una de las enzimas más importantes de la tierra». El trabajo de su equipo se centra en la parte rotatoria de la enzima (Fo), que está oculta dentro de la bicapa lipídica.
Para resolver la estructura del dominio Fo y todo el complejo, los investigadores estudiaron la enzima de las mitocondrias de oveja utilizando microscopía crioelectrónica. Y aquí, la ATP sintasa plantea un problema especial: debido a que gira, la ATP sintasa puede detenerse en tres posiciones principales, así como en subestados. “Es muy difícil distinguir entre estas posiciones, atribuyendo una estructura a cada posición que puede tomar la ATP sintasa. Pero logramos resolver esto computacionalmente para construir la primera estructura completa de la enzima ”, agrega Sazanov.
También descubrieron un enchufe especial que puede detener la máquina cuando la célula está muriendo. Este tapón, denominado «poro de transición de permeabilidad», solo puede abrirse mediante un «aparato de gancho» que está conectado a la parte catalítica en F1 y al fondo de la membrana. Las señales de calcio desencadenan un gran cambio conformacional, que tira del gancho y abre el tapón. Los protones se escapan, desestabilizando el rotor y deteniendo la máquina. Suena como un interruptor de parada de emergencia.
Flagelo bacteriano
También se ha descubierto más acerca de un recurso familiar al DI, el flagelo bacteriano. Científicos europeos dirigidos por Vitan Blagotinsek, que también publican en PNAS2, descubrieron que «un interruptor asociado dependiente de ATP vincula el ensamblaje del anillo C flagelar con la expresión génica». Esto se relaciona con el control de cuántos flagelos construye la célula en la superficie. Algunas bacterias tienen una; otros tienen dos o más. Hasta ahora, la forma en que la célula regula el número de flagelos ha sido “poco conocida”.
En la película Unlocking the Mystery of Life [Desenllavando el misterio de la vida], Scott Minnich se refirió a los mecanismos de retroalimentación que controlan cuántas piezas se envían al sitio de construcción. El equipo europeo aclaró uno de esos comentarios. Descubrieron que el secreto para controlar cuántos flagelos se construyen ocurre cuando una parte impulsada por ATP llamada FlhG activa un interruptor molecular entre otros dos componentes:
Dependiendo de su estado de oligomerización dependiente de ATP, FlhG interactúa con la proteína FliM del anillo C durante el ensamblaje flagelar o con el regulador maestro flagelar FlrA. Este cambio de proteína entre FliM y FlrA establece una red reguladora crítica para la regulación numérica de los flagelos, en la que el ensamblaje físico del flagelo se retroalimenta transcripcionalmente para evitar la producción de más bloques de construcción.
También se obtuvo nueva información sobre el casquete en forma de pentágono que controla la adición ordenada de monómeros a la hélice en crecimiento. Al publicar en Nature Communications3, Al-Otaibi y otras seis personas en el Reino Unido utilizaron microscopía crioelectrónica para estudiar la «estructura del complejo de la tapa del flagelo bacteriano» que «sugiere un mecanismo molecular para el alargamiento del filamento».
La tapa pentagonal, que se parece un poco a las heces, está compuesta de proteínas FliD. Esta tapa se extruye primero en trozos a través del canal central del gancho. Sus subunidades se autoensamblan al final, mientras forman «un extenso conjunto de contactos entre varias subunidades, que contribuyen a la oligomerización de FliD». Luego, la tapa guía a las proteínas de flagelina individuales que suben a través del canal a sus posiciones en el filamento en crecimiento. Descubrieron que se trata de un proceso bien controlado que depende de la disposición precisa de los aminoácidos en cada proteína, de modo que los contactos funcionen según lo previsto.
Una vez fuera de FliD, proponemos que los residuos hidrófobos expuestos actúen como acompañantes y promuevan el plegamiento de flagelina en su sitio de inserción. Para acomodar la siguiente subunidad de flagelina, es necesario que ocurran cambios conformacionales para abrir un bolsillo de unión adyacente. Proponemos que el plegado del nuevo protómero de flagelina conduce al desalojo del complejo del casquete, que gira ~ 65 °, colocando así una cavidad adyacente del complejo del casquete cerca del siguiente sitio de inserción de flagelina (Fig. 5). Esta hipótesis concuerda con los mecanismos de elongación flagelar propuestos previamente.
En cuanto a la especificidad de esta operación, los científicos dicen que «interrumpir estas interacciones tiene un efecto significativo sobre la motilidad celular». Uno recuerda las palabras de Michael Behe: “Si falta alguna de estas partes, entonces o obtiene un flagelo que no funciona, porque le falta el gancho o le falta el eje de transmisión o lo que sea, o ni siquiera se construye dentro de la célula».
Los defensores del DI deben sentirse alentados a ver argumentos a favor de la complejidad irreducible que resisten la prueba del tiempo. Después de más de dos décadas, los evolucionistas todavía «no han podido ofrecer ninguna explicación darwiniana detallada» para un motor rotatorio que surge por selección natural, como lo demuestra el completo silencio de los tres artículos sobre esa cuestión clave.
Notas
- Spikes, Montgomery y Walker, «Estructura de la ATP sintasa dimérica de la mitocondria bovina». PNAS 8 de septiembre de 2020. https://doi.org/10.1073/pnas.2013998117.
- Blagotinsek et al., «Un interruptor de pareja dependiente de ATP vincula el ensamblaje del anillo C flagelar con la expresión génica». PNAS 25 de agosto de 2020117 (34) 20826-20835. https://doi.org/10.1073/pnas.2006470117.
- Al-Otaibi et al., «La estructura crio-EM del complejo de la tapa del flagelo bacteriano sugiere un mecanismo molecular para el alargamiento del filamento». Nature Communications 25 de junio de 2020: 11, 3210 (2020). https://doi.org/10.1038/s41467-020-16981-4.
Artículo originalmente publicado en inglés por Evolution News & Science Today