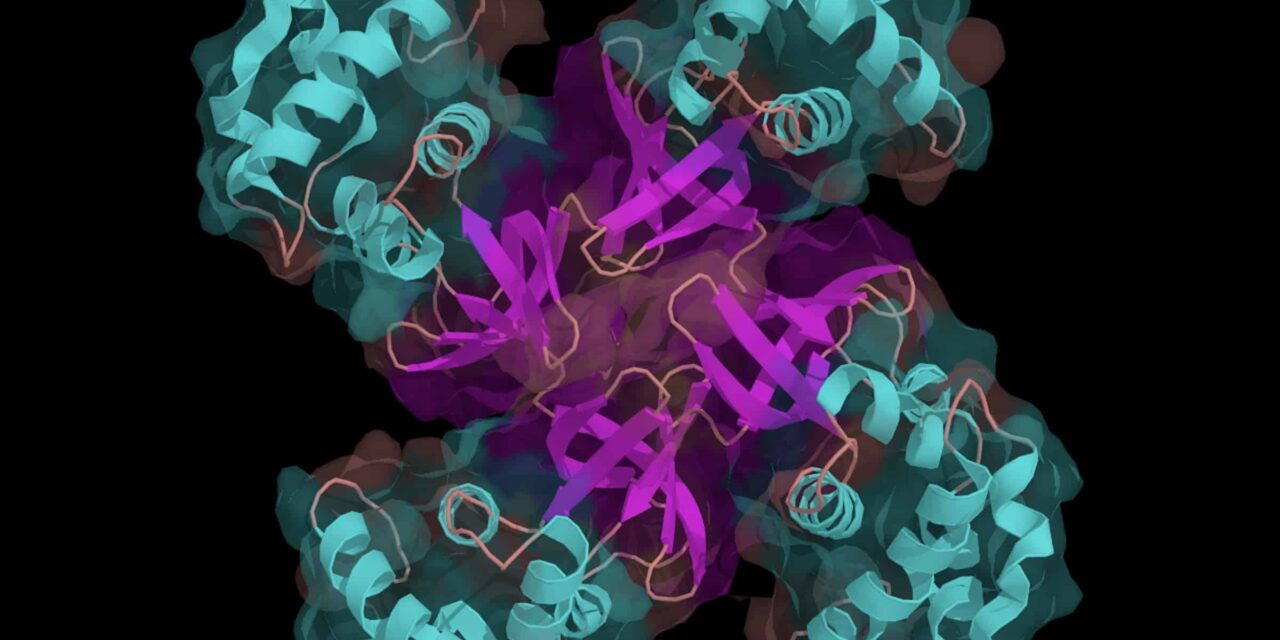
El replisoma de ADN es una de las máquinas moleculares más notables, que involucra un complejo de diferentes proteínas, cada una de las cuales está diseñada de manera muy específica para cumplir su papel en el proceso de replicación del genoma en preparación para la división celular. La tasa de replicación del ADN se ha medido en la sorprendente cantidad de 749 nucleótidos por segundo1 y se cree que la tasa de error para polimerasas precisas está en el rango de 10-7 y 10-8, según estudios de replicación del ADN de E. coli y bacteriófagos. 2
Una de las mejores animaciones de este increíble proceso es esta del animador australiano Drew Berry:
Es difícil mirar una animación como esta (que está drásticamente simplificada) y no tener la fuerte intuición de que una máquina con una coreografía tan intrincada es producto de una ingeniería magistral. Las estructuras proteicas estables y funcionales son astronómicamente raras en el espacio de secuencias combinatorias, y la replicación del ADN requiere muchas de ellas. Pero no cualquier proteína antigua que se pueda plegar de forma estable servirá. Estas proteínas deben elaborarse de manera muy particular para poder realizar sus respectivas funciones. De hecho, cuando uno se centra en proteínas específicas, la intuición del diseño se lleva a nuevas alturas. Por ejemplo, vea estas hermosas animaciones de topoisomerasa, helicasa y ADN polimerasa. Un artículo resume así la destreza de ingeniería de la replicación del ADN:
La síntesis de todo el ADN genómico implica la acción altamente coordinada de múltiples polipéptidos. Estas proteínas ensamblan dos nuevas cadenas de ADN a un ritmo notable, acercándose a los 1.000 nucleótidos (nt) por segundo en E. coli. Si el dúplex de ADN tuviera 1 m de diámetro, entonces las siguientes afirmaciones describirían aproximadamente la replicación de E. coli. La bifurcación se movería a aproximadamente 600 km/h (375 mph) y la maquinaria de replicación sería aproximadamente del tamaño de un camión de reparto de FedEx. Replicar el genoma de E. coli sería un viaje de 40 minutos y 400 km (250 millas) para dos máquinas de este tipo, que, en promedio, cometerían un error sólo una vez cada 170 km (106 millas). La destreza mecánica de este complejo es aún más impresionante dado que sintetiza dos cadenas simultáneamente mientras se mueve. Aunque una hebra se sintetiza en la misma dirección en la que se mueve la horquilla, la otra cadena (la hebra retrasada) se sintetiza poco a poco (como fragmentos de Okazaki) y en la dirección opuesta al movimiento general de la horquilla. Como resultado, aproximadamente una vez una segunda persona de entrega (es decir, el sitio activo de la polimerasa) asociada con el camión debe tomar un desvío, saliendo y luego reuniéndose con su cadena de ADN modelo, para sintetizar los fragmentos de 0,2 km (0,13 millas).3
Complejidad irreducible con esteroides
La replicación del ADN es un ejemplo de lo que podríamos llamar «complejidad irreducible con esteroides». La duplicación del genoma es un requisito previo de la supervivencia diferencial, que es necesaria para que el proceso de selección natural funcione. Por tanto, difícilmente se puede apelar a la selección natural para explicar los orígenes de la replicación del ADN sin asumir la existencia misma de lo que se intenta explicar. Es difícil imaginar un sistema de replicación viable que sea más simple que el replisoma de ADN que se muestra en la animación anterior. Aunque el escenario del mundo del ARN (que sostiene que la vida basada en el ARN es anterior a la vida basada en el ADN y las proteínas) es una hipótesis popular, abundan los problemas para este escenario, como se ha discutido muchas veces aquí y en varias otras publicaciones (por ejemplo, Libro de Stephen Meyer, Signature in the Cell, Capítulo 14). Por ejemplo, uno de los principales desafíos es la inestabilidad inherente del ARN (al ser monocatenario y poseer un grupo 2′ OH adicional, lo que lo hace propenso a la hidrólisis). Por lo tanto, es extremadamente improbable que los polímeros de ARN hayan sobrevivido en el entorno primitivo de la Tierra durante el tiempo suficiente para tener mucho valor. En segundo lugar, cuando el ARN forma pares de bases complementarias para plegarse sobre sí mismo, parte de la molécula ya no presenta una hebra expuesta que pueda servir como plantilla para la copia. Por tanto, existe una limitación física en la capacidad del ARN para autorreplicarse.
Otra razón por la que la maquinaria de replicación del ADN exhibe una complejidad irreductible con esteroides es que, al ser tan primitiva, es mucho más difícil imaginar cualquier tipo de escenario de cooptación que lo sería para un sistema que surgió mucho más tarde, como los flagelos bacterianos. Con el flagelo, al menos se pueden señalar funciones alternativas que podrían realizar varios de los componentes flagelares (como el sistema de secreción tipo III). Sin embargo, con la replicación del ADN, no está claro de qué otros sistemas se podría cooptar cualquiera de los componentes, ya que cualquier otro sistema tendría que haber surgido después de los orígenes de la replicación del ADN.
Un enigma aún más sorprendente es que, en los tres dominios de la vida, las enzimas clave (en particular, las polimerasas replicativas) no son homólogas, lo que ha llevado a sugerir que la replicación del ADN puede haber surgido más de una vez de forma independiente.4,5 Esta observación se adapta más cómodamente a un paradigma de diseño que a uno comprometido con el naturalismo.
¿Qué componentes son esenciales para la replicación del ADN?
¿Qué componentes proteicos que participan en la replicación del ADN son indispensables para su funcionamiento? Primero, está la ADN polimerasa que realmente realiza la copia de cada cadena. Sin él, no se produciría ninguna replicación. Pero la ADN polimerasa no puede comenzar la replicación sin la presencia de un grupo 3′ OH (hidroxilo) libre. Por lo tanto, otra enzima, una forma de ARN polimerasa llamada primasa, crea un fragmento corto de ARN (llamado cebador) desde el cual puede extenderse la ADN polimerasa (a diferencia de la ADN polimerasa, la primasa no requiere la presencia de un grupo 3′ OH libre). ). Por lo tanto, en ausencia de la enzima primasa, no se establecerían cebadores de ARN ni en la cadena principal ni en la cadena retrasada, y la replicación del ADN no podría comenzar. Además, la propia ADN polimerasa debe estar unida al ADN mediante una proteína en forma de anillo conocida como abrazadera deslizante (que evita que se caiga de la cadena plantilla de ADN). Pero la abrazadera deslizante no puede unirse directamente al ADN por sí sola. En cambio, un complejo proteico llamado cargador de abrazadera media la carga de la abrazadera deslizante sobre el ADN en la horquilla de replicación, utilizando la energía de la hidrólisis del ATP para abrir el anillo de la abrazadera deslizante y cargarlo en el ADN. En ausencia de la abrazadera deslizante o del cargador de abrazadera, la ADN polimerasa frecuentemente se desprendería de la plantilla de ADN, volviéndola extremadamente ineficiente.
Por supuesto, el proceso de replicación no puede comenzar a menos que se descomprima la doble hélice del ADN, y esto lo logra la enzima helicasa, que rompe los enlaces de hidrógeno a lo largo de la molécula de ADN, abriendo y exponiendo así las dos hebras para la replicación por la polimerasa. En su ausencia, la ADN polimerasa se detendrá, incapaz de separar las hebras que quedan por delante.
Incluso con la enzima helicasa separando las dos hebras, es probable que las hebras se vuelvan a hibridar durante el proceso de copia. Ingrese a las proteínas de unión monocatenarias que se unen a las cadenas de ADN expuestas, evitando que se vuelvan a hibridar durante la copia. Sin ellos, las cadenas de ADN se volverían a unir antes de poder copiarlas.
Las enzimas topoisomerasas son necesarias para eliminar los superenrollamientos inducidos por la tensión de torsión. Lo hacen cortando un hilo, pasando el otro hilo a través del espacio y luego volviendo a sellar la rotura. En ausencia de las enzimas topoisomerasas, el ADN eventualmente se rompería, dificultando así el proceso de replicación del ADN.
Debido a la naturaleza antiparalela del ADN (y al hecho de que la ADN polimerasa sólo puede replicarse en dirección 5′ a 3′), una cadena, la cadena retrasada, tiene que replicarse hacia atrás (para que la bifurcación de replicación se pueda replicar hacia atrás y moverse en una sola dirección). Esto se hace de forma discontinua en pequeñas secciones. Los cebadores de ARN son fijados por primasa y a partir de ellos se sintetizan fragmentos cortos de ADN conocidos como fragmentos de Okazaki. Luego, los cebadores de ARN se eliminan y se reemplazan con ADN, y la enzima ligasa une los fragmentos de Okazaki. Ya hemos comentado la necesidad de la enzima primasa para sintetizar cebadores de ARN. Se puede agregar que, en ausencia de las enzimas de escisión de ARN (que eliminan los cebadores de ARN), los fragmentos de ARN permanecerían unidos covalentemente a los fragmentos de ADN recién replicados. Además, en ausencia de ligasa (que une los fragmentos de Okazaki), las hebras recién replicadas permanecerían como fragmentos.
Si la eliminación de cualquiera de los componentes antes mencionados haría que la maquinaria de replicación del ADN dejara de funcionar, ¿cómo podría surgir tal sistema a través de una vía darwiniana gradual no dirigida, preservando la utilidad selectiva en cada paso del camino? Cualquiera que fuera el proceso que produjo el ADN replicante, tenía que saber dónde estaba el objetivo. Semejante causa tendría que ser de naturaleza teleológica.
Un paradigma de diseño
La maquinaria de replicación del ADN representa uno de los ejemplos más extraordinarios de nanotecnología encontrados en la célula. En cualquier otro ámbito de la experiencia, una disposición tan compleja y delicada de partes sería inmediatamente reconocida como reflejo de una intención consciente, es decir, como producto de una mente. ¿Por qué debería rechazarse tal inferencia cuando se examinan sistemas biológicos?
Notas
- McCarthy D, Minner C, Bernstein H, Bernstein C. DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant. J Mol Biol. 1976 Oct 5;106(4):963-81.
- Schaaper RM. Base selection, proofreading, and mismatch repair during DNA replication in Escherichia coli. J Biol Chem.1993 Nov 15;268(32):23762-5.
- Baker TA, Bell SP. Polymerases and the replisome: machines within machines. Cell. 1998 Feb 6;92(3):295-305.
- Leipe DD, Aravind L, Koonin EV. Did DNA replication evolve twice independently? Nucleic Acids Res. 1999 Sep 1;27(17):3389-401.
- Brown JR, Doolittle WF. Archaea and the prokaryote-to-eukaryote transition. Microbiol Mol Biol Rev. 1997 Dec;61(4):456-502.